jessD
June 11, 2021, 5:43pm
1
Hi,
I am trying to preprocess the HCP dataset using fMRIPrep. I started by running fMRIPrep using the “IntendedFor” fields to pass the fmaps. The results looked a bit strange.
So, I moved on to run sdcflows to correct the EPI image before applying it to fMRIPrep. I figured out how to use sdcflows pepolar by combining the following worflows (init_topup_wf, fmap_reports_wf, fmap_derivatives_wf. My script looks very similar to this:sdcflows/test_pepolar.py at master · nipreps/sdcflows · GitHub ) and I am creating a pipeline that looks like this:
sdcflows works and it generates the fieldmap, fmap_coeff and fmap_ref files. However, how do I apply the field coefficient image that I got from sdcflow to the EPI image in order to correct it?
jessD
June 24, 2021, 7:27pm
2
I was told that @mgxd is an expert in sdcflows, so I am tagging you here
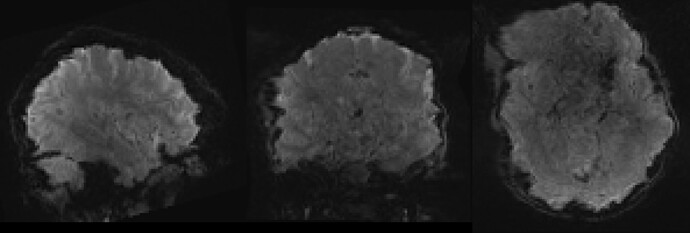
After some guide (thanks @effigies for the help), I was pointed toward this function that uses topup on the sdcflow (sdcflows/test_correction.py at a3765456544b27ed653911b601a14603529beca5 · nipreps/sdcflows · GitHub ). I adapted the test scripts from the sdcflow repo (both scripts with my adaptations to run on a test subject can be found here GitHub - JessyD/sdcflows-test ). After running the apply_correction.py I got the following outputs:
$ tree test_out/
test_out/
└── sdcflows
└── sub-132118
└── figures
├── sub-132118_task-rest_acq-rl_run-1_desc-corrected_bold.svg
└── sub-132118_task-rest_acq-rl_run-1_desc-fieldmap_bold.svg
$ tree test_wk
test_wk
└── test_unwarp_wf
├── coeff2epi_wf
│ ├── coregister
│ │ ├── _0xefefcf3bbafeddc37e0d45724876090d.json
│ │ ├── command.txt
│ │ ├── _inputs.pklz
│ │ ├── _node.pklz
│ │ ├── _report
│ │ │ └── report.rst
│ │ ├── result_coregister.pklz
│ │ ├── transform0GenericAffine.mat
│ │ └── transform_Warped.nii.gz
│ └── map_coeff
│ ├── _0xba48cd7ba0e71044993b420e0c71f5f8.json
│ ├── _inputs.pklz
│ ├── moved_coeff_000.nii.gz
│ ├── _node.pklz
│ ├── _report
│ │ └── report.rst
│ └── result_map_coeff.pklz
├── d3.js
├── ds_fmap_report
│ ├── _0xf42168cee2396d86b7ba114284dd9fc3.json
│ ├── _inputs.pklz
│ ├── _node.pklz
│ ├── _report
│ │ └── report.rst
│ └── result_ds_fmap_report.pklz
├── ds_report
│ ├── _0xb8dcee9196100183bf1c721e3f0cdd3d.json
│ ├── _inputs.pklz
│ ├── _node.pklz
│ ├── _report
│ │ └── report.rst
│ └── result_ds_report.pklz
├── epi_ref_wf
│ ├── brainextraction_wf
│ │ ├── clipper_post
│ │ │ ├── _0x171e81e1ca311b88baef7927009a7a1e.json
│ │ │ ├── clipped.nii.gz
│ │ │ ├── _inputs.pklz
│ │ │ ├── _node.pklz
│ │ │ ├── _report
│ │ │ │ └── report.rst
│ │ │ └── result_clipper_post.pklz
│ │ ├── clipper_pre
│ │ │ ├── _0xe98837389469c395b65acd7014e60712.json
│ │ │ ├── clipped.nii.gz
│ │ │ ├── _inputs.pklz
│ │ │ ├── _node.pklz
│ │ │ ├── _report
│ │ │ │ └── report.rst
│ │ │ └── result_clipper_pre.pklz
│ │ ├── masker
│ │ │ ├── _0xffe8c6be272448662ddd6b0c50d38304.json
│ │ │ ├── clipped_brainmasked.nii.gz
│ │ │ ├── clipped_mask.nii.gz
│ │ │ ├── clipped_probseg.nii.gz
│ │ │ ├── _inputs.pklz
│ │ │ ├── _node.pklz
│ │ │ ├── _report
│ │ │ │ └── report.rst
│ │ │ └── result_masker.pklz
│ │ └── n4
│ │ ├── _0x849dcab6ede3f1bd4a6867e9f3c766cd.json
│ │ ├── clipped_corrected.nii.gz
│ │ ├── command.txt
│ │ ├── _inputs.pklz
│ │ ├── _node.pklz
│ │ ├── _report
│ │ │ └── report.rst
│ │ └── result_n4.pklz
│ └── magmrg
│ ├── _0x8cf8f0b1354a805ccf09b9d84e77b326.json
│ ├── _inputs.pklz
│ ├── _node.pklz
│ ├── _report
│ │ └── report.rst
│ ├── result_magmrg.pklz
│ └── sub-132118_task-rest_acq-rl_run-01_bold_avg.nii.gz
├── fmap_ref_wf
│ ├── brainextraction_wf
│ │ ├── clipper_post
│ │ │ ├── _0x050d7d48fedb06f78fe6224559601163.json
│ │ │ ├── clipped.nii.gz
│ │ │ ├── _inputs.pklz
│ │ │ ├── _node.pklz
│ │ │ ├── _report
│ │ │ │ └── report.rst
│ │ │ └── result_clipper_post.pklz
│ │ ├── clipper_pre
│ │ │ ├── _0x5e0e53f7a554b668426c4f1b71ead26c.json
│ │ │ ├── clipped.nii.gz
│ │ │ ├── _inputs.pklz
│ │ │ ├── _node.pklz
│ │ │ ├── _report
│ │ │ │ └── report.rst
│ │ │ └── result_clipper_pre.pklz
│ │ ├── masker
│ │ │ ├── _0x74f5e07128e8f27410eeaf720fd593d2.json
│ │ │ ├── clipped_brainmasked.nii.gz
│ │ │ ├── clipped_mask.nii.gz
│ │ │ ├── clipped_probseg.nii.gz
│ │ │ ├── _inputs.pklz
│ │ │ ├── _node.pklz
│ │ │ ├── _report
│ │ │ │ └── report.rst
│ │ │ └── result_masker.pklz
│ │ └── n4
│ │ ├── _0x13930d6f967a3a5c3a6aba16b3e84a8c.json
│ │ ├── clipped_corrected.nii.gz
│ │ ├── command.txt
│ │ ├── _inputs.pklz
│ │ ├── _node.pklz
│ │ ├── _report
│ │ │ └── report.rst
│ │ └── result_n4.pklz
│ └── magmrg
│ ├── _0x4899c4151f900a4db31d42a195dbbeda.json
│ ├── _inputs.pklz
│ ├── _node.pklz
│ ├── _report
│ │ └── report.rst
│ ├── result_magmrg.pklz
│ └── sub-132118_run-01_magnitude2_merged.nii.gz
├── graph1.json
├── graph_detailed.dot
├── graph_detailed.png
├── graph.dot
├── graph.json
├── graph.png
├── index.html
├── report
│ ├── _0xb4b88707e190204003a3da058a6c1b06.json
│ ├── _inputs.pklz
│ ├── _node.pklz
│ ├── _report
│ │ └── report.rst
│ ├── report.svg
│ └── result_report.pklz
├── simple_report
│ ├── _0x91d54b913e8d0a385d2fbdb9ff0d6214.json
│ ├── _inputs.pklz
│ ├── _node.pklz
│ ├── _report
│ │ └── report.rst
│ ├── report.svg
│ └── result_simple_report.pklz
└── unwarp_wf
├── brainextraction_wf
│ ├── clipper_post
│ │ ├── _0xe57212b5964b8675708ad84225497c95.json
│ │ ├── clipped.nii.gz
│ │ ├── _inputs.pklz
│ │ ├── _node.pklz
│ │ ├── _report
│ │ │ └── report.rst
│ │ └── result_clipper_post.pklz
│ ├── clipper_pre
│ │ ├── _0x5a8ac87ed7d006f6dc8884764fb469b5.json
│ │ ├── clipped.nii.gz
│ │ ├── _inputs.pklz
│ │ ├── _node.pklz
│ │ ├── _report
│ │ │ └── report.rst
│ │ └── result_clipper_pre.pklz
│ ├── masker
│ │ ├── _0x9c079a06b45cd8a3607ad666cd68d604.json
│ │ ├── clipped_brainmasked.nii.gz
│ │ ├── clipped_mask.nii.gz
│ │ ├── clipped_probseg.nii.gz
│ │ ├── _inputs.pklz
│ │ ├── _node.pklz
│ │ ├── _report
│ │ │ └── report.rst
│ │ └── result_masker.pklz
│ └── n4
│ ├── _0x1cf1f0f69d0e211ea13d8d17a3224169.json
│ ├── clipped_corrected.nii.gz
│ ├── command.txt
│ ├── _inputs.pklz
│ ├── _node.pklz
│ ├── _report
│ │ └── report.rst
│ └── result_n4.pklz
├── resample
│ ├── _0xc8cbd63152fed8b14bd2b8320cbe855c.json
│ ├── clipped_unwarped.nii.gz
│ ├── clipped_xfm.nii.gz
│ ├── _inputs.pklz
│ ├── moved_coeff_000_field.nii.gz
│ ├── _node.pklz
│ ├── _report
│ │ └── report.rst
│ └── result_resample.pklz
└── rotime
├── _0x0303e24b0aea322e120c5207631d6949.json
├── _inputs.pklz
├── _node.pklz
├── _report
│ └── report.rst
└── result_rotime.pklz
However, all of the images in the working directory contain just one single volume, so I am still not sure how to apply the transformation to my EPI image. Is there another function that I am missing that will take the output from the topup workflow and apply it to an EPI image?